| 일 | 월 | 화 | 수 | 목 | 금 | 토 |
|---|---|---|---|---|---|---|
| 1 | 2 | 3 | ||||
| 4 | 5 | 6 | 7 | 8 | 9 | 10 |
| 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| 18 | 19 | 20 | 21 | 22 | 23 | 24 |
| 25 | 26 | 27 | 28 | 29 | 30 | 31 |
- MySQL
- hackerrank
- 파이썬
- 스파르타코딩
- 파이썬 머신러닝 완벽가이드
- 실전 데이터 분석 프로젝트
- 오블완
- Cluster
- SQL
- 프로젝트
- 미세먼지
- 스파르타
- 텍스트 분석
- 프로그래머스
- 파이썬 철저 입문
- R
- 내일배움카드
- 내일배움캠프
- 스파르타 코딩
- 파이썬 완벽 가이드
- harkerrank
- 파이썬 철저입문
- 파이썬 머신러닝 완벽 가이드
- wil
- 웹 스크랩핑
- TiL
- 티스토리챌린지
- 내일배움
- 회귀분석
- 중회귀모형
- Today
- Total
OkBublewrap
로지스틱 회귀분석 본문
지도학습 : 분류, 회귀
유방암데이터
import pandas as pd
import numpy as np
# 시각화
import matplotlib.pyplot as plt
%matplotlib inline
# 유방암 데이터
from sklearn.datasets import load_breast_cancer
# 로지스틱회귀분석 모델
from sklearn.linear_model import LogisticRegression
cancer = load_breast_cancer()
type(cancer)
# sklearn.utils.Bunch
cancer.keys()
# dict_keys(['data', 'target', 'frame', 'target_names', 'DESCR', 'feature_names', 'filename', 'data_module'])
# 데이터프레임 만들기
cancer_df = pd.DataFrame(data=cancer.data, columns=cancer.feature_names)
cancer_df['target'] = cancer.target
np.bincount(cancer.target)
# array([212, 357])0 : 악성 종양, 1 : 악성이 아닌 종양
악성 종양은 212, 악성이 아닌 종양은 357인 target의 분포이다.
Columns :
'mean radius', 'mean texture', 'mean perimeter', 'mean area', 'mean smoothness', 'mean compactness', 'mean concavity', 'mean concave points', 'mean symmetry', 'mean fractal dimension', 'radius error', 'texture error', 'perimeter error', 'area error', 'smoothness error', 'compactness error', 'concavity error', 'concave points error', 'symmetry error', 'fractal dimension error', 'worst radius', 'worst texture', 'worst perimeter', 'worst area', 'worst smoothness', 'worst compactness', 'worst concavity', 'worst concave points', 'worst symmetry', 'worst fractal dimension', 'target'
# 데이터 전처리(평균 0, 분산 1)
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
# x
data_scaled = scaler.fit_transform(cancer.data)
# 데이터 분할(7:3)
from sklearn.model_selection import train_test_split
X_train , X_test, y_train , y_test = train_test_split(data_scaled, cancer.target,
test_size=0.3, random_state=0)로지스틱 회귀
# 시그모이드 함수
import matplotlib.pyplot as plt
import numpy as np
def sigmoid(x):
y = 1/(1 + np.exp(-x))
return y
x = np.arange(-7, 7, 0.1)
plt.plot(x, sigmoid(x))
plt.axvline(0, color='k')
plt.axhline(0.5, color='k')
plt.show()
로지스틱 함수
$$ p(x)=1/(1+e^{-x})=e^{x}/(1+e^{x}) $$
오즈
$$ odds = \left ( \frac{p}{1-p} \right ) $$
로짓 함수
$$ logit(p) = log\left ( \frac{p}{1-p} \right ), p \in (0,1) $$
오즈비
$$ R = \left ( \frac{p_{1}/(1-p_{1})}{p_{2}/(1-p_{2})} \right ) $$
$$ log(R) = \left ( \frac{p_{1}/(1-p_{1})}{p_{2}/(1-p_{2})} \right ) = log\left ( \frac{p_{1}}{1-p_{1}} \right )-log\left ( \frac{p_{2}}{1-p_{2}} \right )= logit(p_{1})-logit(p_{2}) $$
# Modeling
lr_clf = LogisticRegression()
lr_clf.fit(X_train, y_train)
# LogisticRegression으로 예측한 값
lr_pred = lr_clf.predict(X_test)
# 실제값
# y_test
# 혼동행렬
from sklearn.metrics import confusion_matrix
import seaborn as sns
cm = confusion_matrix(y_test,lr_preds)
cm = pd.DataFrame(cm, index=['실제값(N)', '실제값(P)'], columns=['예측값(N)', '예측값(P)'])
sns.heatmap(cm, annot = True, fmt = 'd',cmap = 'Reds')
plt.xlabel('True')
plt.ylabel('pred')
plt.xticks([0.5,1.5],['0(N)', '1(P)'])
plt.yticks([0.5,1.5],['0(N)', '1(P)'])
plt.show()
from sklearn.metrics import accuracy_score, roc_auc_score, f1_score, precision_score, recall_score
print('accuracy: {:0.3f}'.format(accuracy_score(y_test, lr_preds)))
print('roc_auc: {:0.3f}'.format(roc_auc_score(y_test , lr_preds)))
print('f1: {:0.3f}'.format(f1_score(y_test, lr_preds)))
print('precision: {:0.3f}'.format(precision_score(y_test, lr_preds)))
print('recall: {:0.3f}'.format(recall_score(y_test, lr_preds)))accuracy: 0.977 roc_auc: 0.972 f1: 0.982 precision: 0.973 recall: 0.991
대체로 score은 좋게 나온것을 확인할 수 있다.
# 최적의 파라미터 구하기(규제)
from sklearn.model_selection import GridSearchCV
# 파라미터값 지정
params={'penalty':['l2', 'l1'], 'C':[0.01, 0.1, 1, 1, 5, 10]}
lr_clf2 = LogisticRegression(solver = "liblinear")
grid_clf2 = GridSearchCV(lr_clf2, param_grid = params, scoring = 'accuracy', cv = 3)
grid_clf2.fit(data_scaled, cancer.target)
print('최적 하이퍼 파라미터:{0}, 최적 평균 정확도:{1:.3f}'.format(grid_clf2.best_params_,
grid_clf2.best_score_))최적 하이퍼 파라미터:{'C': 0.1, 'penalty': 'l2'}, 최적 평균 정확도:0.979다중 로지스틱회귀(softmax regression)
# iris data
from sklearn import datasets
iris = datasets.load_iris()
# train_test_spilt
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(iris.data, iris.target, test_size = 0.3, random=state = 2022)
# model
from sklearn.liner_model import LogisticRegression
softmax_reg = LogisticRegression(mult_class = "multinomial")
softmax_reg.fit(X_train, y_train)
pred = softmax_reg.predict(X_test)
# 실제값, 예측값, 예측확률 데이터프레임
mult_reg = pd.DataFrame({'true' : y_test, 'pred': pred})
mult_reg2 = pd.concat([mult_reg, pd.DataFrame(pred_prob)], axis = 1)
mult_reg2.head(5)
예측확률 값을 보면 0번째는 2의 확률이 0.9으로 나온것을 확인할 수 있다. 전반적으로 잘 예측 한것을 볼 수 있다.
# train score
softmax_reg.score(X_train, y_train)
# 0.9809523809523809
# test score
softmax_reg.score(X_test, y_test)
# 0.9777777777777777train에서는 0.98의 정확도를 보여주고 있다. 반면 test에서는 0.97의 정확도를 보여주고 있다.
mult_reg2[mult_reg2['true'] != mult_reg2['pred']]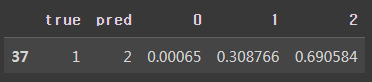
규제 C = 0.1
softmax_reg = LogisticRegression(multi_class = "multinomial", C = 0.1)
softmax_reg.fit(X_train, y_train)
pred = softmax_reg.predict(X_test)
pred_prob = softmax_reg.predict_proba(X_test)
softmax_reg.score(X_test, y_test)
# 0.9333333333333333규제 C = 10
softmax_reg = LogisticRegression(multi_class = "multinomial", C = 10)
softmax_reg.fit(X_train, y_train)
pred = softmax_reg.predict(X_test)
pred_prob = softmax_reg.predict_proba(X_test)
softmax_reg.score(X_test, y_test)
# 0.9777777777777777c = 0.1 일때는 score 0.933, 10일 때는 0.97으로 나왔다. 성능이 조금 더 개선 된 것을 확인할 수 있다.
회귀 트리
from sklearn.datasets import load_boston
from sklearn.model_selection import cross_val_score
from sklearn.model_selection import train_test_split
from sklearn.tree import DecisionTreeRegressor
import pandas as pd
import numpy as np
# 데이터 불러오기
boston = load_boston()
bostonDF = pd.DataFrame(boston.data, columns = boston.feature_names)
# 집 값이 타겟 변수
bostonDF['PRICE'] = boston.target
y_target = bostonDF['PRICE']
# 예측 변수에 집 값 삭제
X_data = bostonDF.drop(['PRICE'], axis=1,inplace=False)
# 데이터 나누기
X_train , X_test, y_train , y_test = train_test_split(X_data, y_target,
test_size=0.3, random_state=0)
# 회귀나무 만들기(최대 나무깊이 2)
tree_reg = DecisionTreeRegressor(max_depth=2, random_state=0)
tree_reg.fit(X_train, y_train)
pred = tree_reg.predict(X_test)
tree_reg.score(X_test, y_test)
# 0.622596538377147
# Graphviz 시각화
import graphviz
from sklearn.tree import export_graphviz
export_graphviz(tree_reg, out_file ="tree.dot", feature_names=boston.feature_names, filled=True)
with open("tree.dot") as f:
dot_graph = f.read()
graphviz.Source(dot_graph)
회귀트리 score 값은 0.622으로 나왔다.
# 랜덤포레스트
from sklearn.ensemble import RandomForestRegressor
rf_reg = RandomForestRegressor(random_state=0, n_estimators=1000)
rf_reg.fit(X_train, y_train)
pred = rf_reg.predict(X_test)
rf_reg.score(X_test, y_test)
# 0.8286153119401956랜덤포레스트 값은 0.828으로 회귀트리보다 더 좋은 성능을 보여주고 있다.
# seaborn 시각화
import seaborn as sns
%matplotlib inline
rf_reg.fit(X_train, y_train)
# 변수 중요도
feature_series = pd.Series(data=rf_reg.feature_importances_, index=X_data.columns )
feature_series = feature_series.sort_values(ascending=False)
sns.barplot(x= feature_series, y=feature_series.index)
랜덤 포레스트 변수 중요도를 시각화 한 결과는 RM, LSTAT이 가장 중요하게 나왔다.
'Python > 학습용' 카테고리의 다른 글
| LDA(선형판별분석) (1) | 2022.12.06 |
|---|---|
| 주성분분석(PCA) (0) | 2022.12.06 |
| 회귀분석(4) (0) | 2022.11.21 |
| 회귀분석(3) (0) | 2022.11.21 |
| 회귀분석(2) (0) | 2022.11.10 |





